本文讲述医学图像配准中形变场的可视化,包括一种直接通过工具查看的方法和两种手工绘制的方法。
首先来介绍一下形变场,一个大小为[W,H]的二维图像对应的形变场的大小是[W,H,2],其中第三个维度的大小为2,分别表示在x轴和y轴方向的位移。同理,一个大小为[D,W,H]的三维图像对应的形变场的大小是[D,W,H,3],其中第三个维度的大小为3,分别表示在x轴、y轴和z轴方向的位移。下图是一个二维脑部图像配准后得到的形变场。

如一开始所说,下面介绍3中方式来将形变场可视化。
1. 使用ITK-Snap可视化形变场
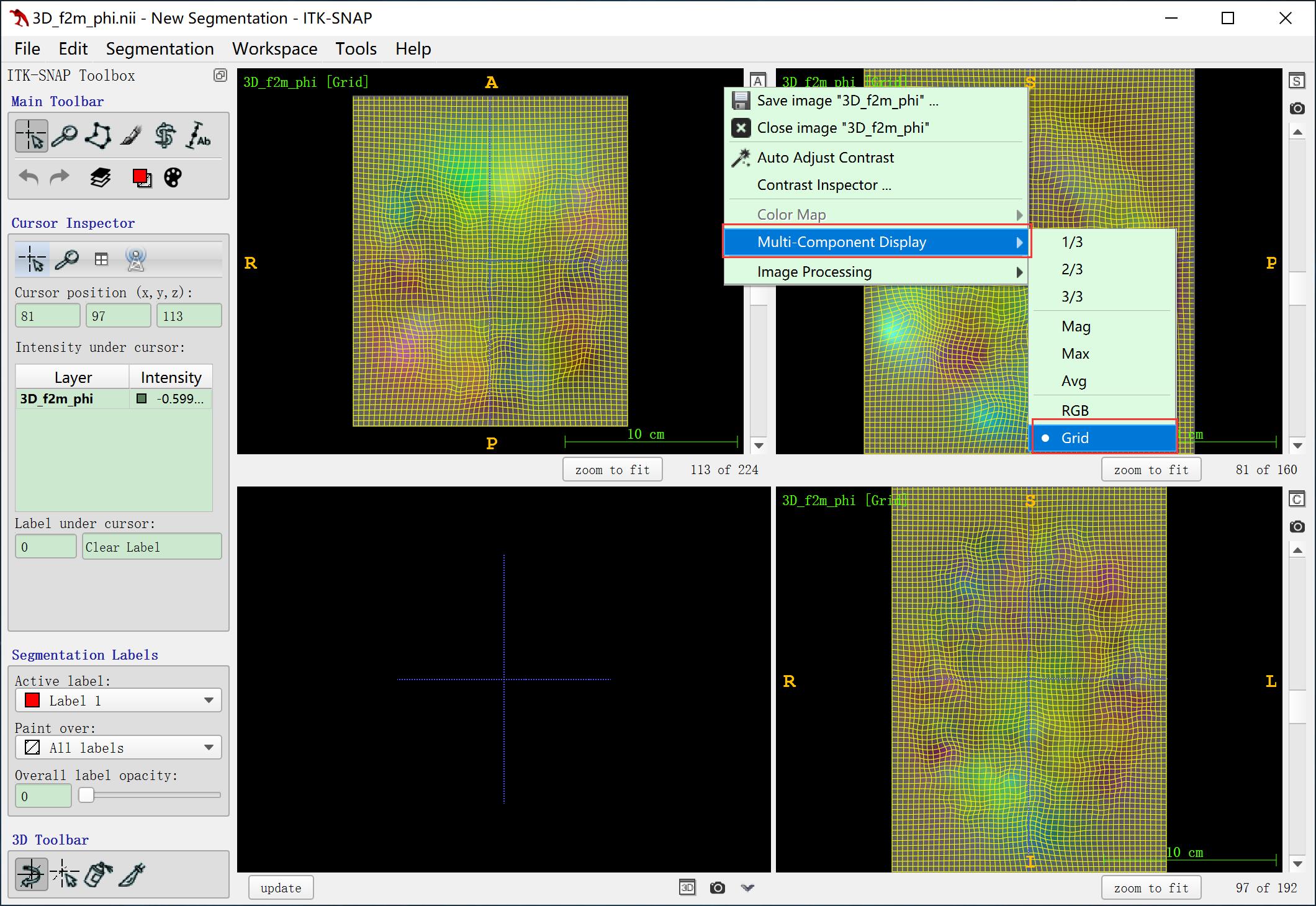
这种方式是最便捷也是我认为效果最好的,但是只适用于三维的形变场。首先将形变场保存为”.nii”图像,用ITK-Snap工具打开,首先在下图红框的位置悬停一段时间,出现下图中的图标后点击,选择“Multi-Component Display”下面的“Grid”,就可以看到下图所示的效果。
2. 毛毛大神的方法
具体思路请看毛毛大神的文章plt.contour 绘制图像形变场(Deformation Field)。其实具体思路我没有完全看懂,大约是先产生一个规则的网格,然后加上形变场后展示出来。但是如果网格是[W,H]大小的,但是形变场是[W,H,2]大小的,所以只能选择一个维度相加,感觉有点奇怪。代码如下:
1 | import matplotlib.pyplot as plt |
在代码的第20行,改变levels的值,可以改变形变场中网格的密集程度。
3. 先生成规则网格,再用空间变换网络进行变形
这种方式是配准群里的一位朋友提出的,自己进行实现。思路是先生成规则网格并保存为图片,再用空间变换网络进行变形。代码如下:
1 | import numpy as np |
需要一提的是,由于不能精确的决定网格图片的大小,所以在生成的时候多生成了10个像素,然后在读取的时候再进行裁剪。改变上述代码中第57行的除数(10)可以改变网格线的密集程度。
感觉这种方式更优雅一点,但是效果和第二种是差不多的,第二种和第三种方法的效果如下图所示:

- 本文作者: 俎志昂
- 本文链接: zuzhiang.cn/2020/07/17/show-phi/
- 版权声明: 本博客所有文章除特别声明外,均采用 Apache License 2.0 许可协议。转载请注明出处!


